コラム
MEA (Multi-Electrode Array:多電極アレイ) アッセイとは
このページでは、ヒトiPS細胞分化細胞を用いたアッセイ技術情報のトピックをご紹介します。
MEAアッセイとは、プレート底面に埋め込まれた微小多点電極を用い、細胞の細胞外活動電位をリアルタイムで測定する電気生理学的試験・評価システムです。主に心筋細胞と神経細胞の測定に用いられています。
私たちは、ヒトiPS細胞由来の神経細胞の測定に用いるMEAアッセイ系を開発し、それを用いたサービスを提供しています。

心筋細胞を用いたMEAアッセイは、心筋細胞の収縮・弛緩の解析、化合物の心毒性スクリーニング、心疾患の表現型解析などに活用されています[1]~[3]。
2023年5月に発表された東北大学らの共同研究では、COVID-19治療薬のレムデシビルが心機能に与える副作用のメカニズム解明にヒトiPS細胞由来の心筋細胞を用いたMEAアッセイが用いられました[4]。レムデシビルが細胞膜のウロテンシン受容体を活性化し、ヒトQT時間に相当する指標を延長すること、ウロテンシン受容体拮抗剤で延長が抑制されることが確認されました。この成果は、レムデシビルの副作用を受容体経路の抑制で改善できる可能性を示しています。

一方、神経細胞を用いたMEAアッセイは、神経電気生理学的特性の解析、化合物の神経毒性スクリーニング、神経疾患の表現型解析などに活用されています[5]~[7]。
神経疾患の候補薬は、臨床試験で毒性が判明して開発中止になることが特に多く、これが大きな問題になっています[8]。一般的に、臨床試験前の毒性スクリーニングには動物実験が行われますが、神経系はヒトと動物で種差が大きいため、正確な結果を得られるとは限りません[9]。
ヒトiPS細胞由来の神経細胞を用いたMEAアッセイは新薬候補の神経毒性スクリーニングの精度を高め、開発期間やコストを削減すると期待されています。

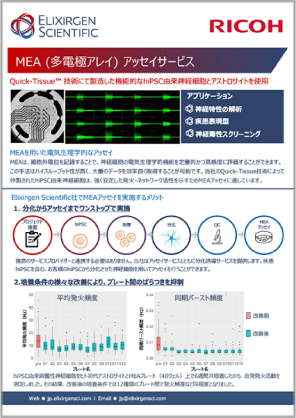
しかし、ヒトiPS細胞由来の神経細胞を用いたMEAアッセイは、長期培養で細胞の凝集や剥がれなどが起こる場合もあり、安定的に行うには特殊なノウハウが必要です。
私たちは、バイオプリンティング開発等で培った材料プロセス技術を応用し、基材への細胞接着を大幅に改善しました。これにより、長期培養でも安定した品質でMEAアッセイが可能になりました。この結果、神経電気生理学的特性の解析や神経毒性スクリーニングの結果を安定して得ることができています。以下の『MEAアッセイ関連技術資料』をご参照ください。
ヒトiPS細胞由来の神経細胞を用いたMEAアッセイは神経科学の分野で有効な解析手法の一つです。リコーは創薬研究へのさらなる貢献を目指して開発を進めてまいります。
MEAアッセイ関連の技術資料
ポスター
-
ポスター1
ASSAY DEVELOPMENT FOR THE CHARACTERIZATION OF SPINE FORMATION AND NETWORK MATURATION OF IPSC-DERIVED NEURONS(ISSCR 2024 国際幹細胞研究学会)
-
ポスター4
Electrophysiological assay system with iPSC-derived neurons and astrocytes generated by a rapid differentiation method (NEURO 2024 日本神経科学大会)
-
ポスター6
多点電極アレイ(MEA)を用いた、自閉症スペクトラムドナーのiPS細胞由来神経細胞の神経活動評価(NEURO 2022 日本神経科学大会)
-
ポスター8
Assay development for the characterization of spine formation and network maturation of iPSC-derived neurons (Society for Neuroscience 2024北米神経科学学会)
-
ポスター9
Electrophysiological assay system based on iPSC-derived astrocytes generated by a rapid differentiation method (Society for Neuroscience 2024北米神経科学学会)
インフォシート
-
インフォシート1
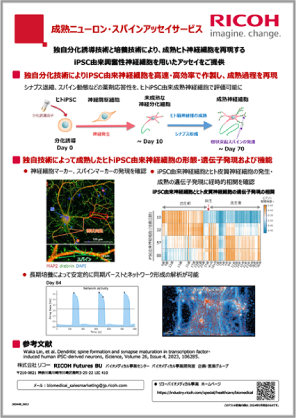
成熟ニューロン・スパインアッセイサービス
リコーグループが提供する、成熟ヒト神経細胞を再現するヒトiPS細胞由来の神経細胞を用いたアッセイサービスの詳細をご紹介しています。
-
インフォシート2
MEAアッセイサービス
リコーグループが提供する、ヒトiPS細胞由来の神経細胞を用いたMEAアッセイサービスの詳細をご紹介してます。
論文
-
2023 2.27Lin W et al . Dendritic spine formation and synapse maturation in transcription factor-induced human iPSC-derived neurons
doi: 10.1016/j.isci.2023.106285
参考文献
- Simeonov S et al. Anal. Chem. 2019;91(15):9648–9655.
doi:10.1021/acs.analchem.9b01092 - Zwartsen A et al. J Mol Cell Cardiol. 2019;136:102-112.
doi:10.1016/j.yjmcc.2019.09.007 - Yang J et al. Front Physiol. 2021;12:778982.
doi:10.3389/fphys.2021.778982 - Ogawa A et al. Commun Biol. 2023;6(1):511.
doi:10.1038/s42003-023-04888-x - Pelkonen A et al. Cells. 2021;11(1):106.
doi:10.3390/cells11010106 - Durens M et al. J Neurosci Methods. 2020;335:108627.
doi:10.1016/j.jneumeth.2020.108627 - McCready FP et al. Biology (Basel). 2022;11(2):316.
doi:10.3390/biology11020316 - Pankevich DE et al. Neuron. 2014;84(3):546-553.
doi:10.1016/j.neuron.2014.10.007 - Van Norman GA. JACC Basic Transl Sci. 2019;4(7):845-854.
doi:10.1016/j.jacbts.2019.10.008